Il CRS4 è stato un pioniere nell’uso del paradigma di programmazione MapReduce per l’elaborazione scalabile dei dati di sequenziamento.
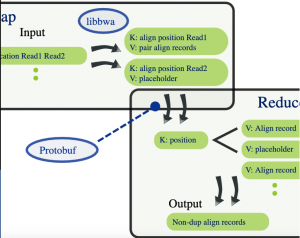
Tra i suoi vari contributi in questo ambito, il CRS4 ha prodotto uno dei primi programmi che hanno utilizzato questo paradigma per calcolare la mappatura della sequenza del DNA e identificare le sequenze duplicate dovute al processo di preparazione del campione biologico (a sinistra).
Per aiutare ad integrare questa sofisticata tecnologia nelle pratiche di laboratorio già esistenti e renderla più accessibile agli utenti finali, il CRS4 ha integrato questi componenti in un sistema web di gestione del workflow ampiamente diffuso, realizzando così una piattaforma di analisi dei dati di facile utilizzo, supportata dalla scalabilità e dalla potenza di calcolo degli strumenti e dell’infrastruttura informatica del CRS4.
L'articolo seguente ha introdotto SEAL, uno dei primi programmi di allineamento di sequenze di DNA basati su Hadoop, insieme ad un toolkit di accompagnamento per l'elaborazione distribuita di sequenze di DNA. È stato il primo di una serie di contributi del CRS4 che mirava a fornire un'elaborazione scalabile delle sequenze su hardware di base, invece di affidarsi a costosi nodi di calcolo di grandi dimensioni e a grandi filesystem cluster-shared.
- L Pireddu, S Leo, G Zanetti. SEAL: a distributed short read mapping and duplicate removal tool Bioinformatics 27 (15), 2011: 2159-2160.